
20
ISSN 2477-9105 Número 22 Vol. 2 (2019)
ESTUDIO DEL METABOLOMA EN EL ANTAGONISMO MICROBIANO
A TRAVÉS DE CROMATOGRAFÍA LÍQUIDA CON ALGORITMOS
QUIMIOMÉTRICOS
R
esumen
El metaboloma es el conjunto de compuestos orgánicos de bajo peso molecular (metabolitos) pro-
ducidos por sistemas biológicos. El antagonismo microbiano es una importante fuerza evolutiva,
por lo que el análisis de su metaboloma es una herramienta útil para la búsqueda de nuevas molé-
culas con actividad biológica. El objetivo de este trabajo fue implementar el uso de algoritmos qui-
miométricos para la identificación de variaciones en el metaboloma del antagonismo microbiano
entre Pseudovibrio denitrificans y Vibrio harveyi. Extractos de bacterias y del medio de cultivo
usado fueron analizados por cromatografía líquida de ultra alta eficiencia acoplada a un detec-
tor de arreglo de diodos (UHPLC-DAD). Además, algoritmos quimiométricos fueron aplicados
realizando un Análisis de Componentes Principales (PCA) de los cromatogramas obtenidos. Se
encontraron tres picos que expresaron una mayor variabilidad entre el metaboloma individual y
el metaboloma de la interacción de P. denitrificans y V. harveyi. De esta manera, la metabolómica
con UHPLC-DAD y algoritmos quimiométricos demostró ser una herramienta útil para la identi-
ficación de picos responsables de las diferencias entre interacciones microbianas.
Palabras Claves: Antagonismo microbiano, cromatografía líquida, análisis de componentes prin-
cipales, Pseudovibrio denitrificans, Vibrio harveyi.
Metabolome analysis in the microbial antagonism by liquid chromatography coupled
with chemometrics algorithms
1
B. Chalén-Alvarado,
2
C. Quiroz-Moreno* ,
2
NGS. Mogollón ,
1
C. Domínguez ,
1
J. Rodríguez
1
Escuela Superior Politécnica del Litoral, ESPOL, Centro Nacional de Acuicultura e Investi-
gaciones Marinas - CENAIM, Guayaquil, Ecuador.
2
Universidad Regional Amazónica Ikiam, Tena, Ecuador
§Ambos autores contribuyeron de forma igualitaria para este artículo
*cristianquirozd1997@gmail.com
A
bstract
Metabolome is a group of low molecular weight organic compounds (metabolites) produced by
biological systems. Bacterial antagonism is an important evolving force, in this sense analysis of
its metabolome represents a useful tool for discovering new molecules with biological activity.
The objective of this research work was to implement the use of chemometric algorithms for the
identification of variations in the metabolome of the microbial antagonism between Pseudovibrio
denitrificans and Vibrio harveyi. Extracts from cultured bacteria and used culture media were
analyzed by Ultra-high performance liquid chromatography coupled with a diode-array detector
(UHPLC-DAD). Additionally, chemometric algorithms were employed, subjecting to Principal
Component Analysis (PCA) for the obtained chromatograms. Three peaks were found that
express the major variability between the individual metabolome and the metabolome from the
interaction of P. denitrificans and V. harveyi. In this manner, metabolomic through UHPLC-
DAD and chemometric algorithms, showed to be a useful tool to identify the peaks responsible for
differences between microbial interactions.
Keywords: Microbial antagonism, liquid chromatography, principal component analysis,
Pseudovibrio denitrificans, Vibrio harveyi.
Fecha de recepción: 30-08-2018 Fecha de aceptación: 17-06-2019

21
Chalén, Quiroz,Mogollon, Domínguez, Rodríguez
I. INTRODUCCIÓN
El antagonismo es un tipo de interacción en-
tre especies en la cual dos o más individuos
tienen una posición de hostilidad o compe-
tencia, ya sea por recursos, territorio o repro-
ducción. Esta presión por competición es una
de las mejores fuerzas evolutivas en micro-
biomas, donde una especie debe asegurar su
supervivencia sobre la enorme diversidad de
microorganismos que la acompañan (1). En
el ámbito marino, encontramos que las inte-
racciones de bacterias asociadas con arreci-
fes de coral intervienen en el mantenimiento
de la salud de este bioma. Entre el amplio
espectro de bacterias asociadas a biomas de
arrecifes se encuentran los géneros Pseudovi-
brio y Vibrio, los cuales comparten hábitat y
presentan altos índices de antagonismo entre
ellos (2). Estudios basados en el antagonismo
microbiano se han usado para desarrollar
biocontroles contra microorganismos per-
judiciales en diversos campos (3–6). La ex-
presión de compuestos activos presentes en
los microbiomas que intervienen en la com-
petición presentan una oportunidad para el
aislamiento de moléculas bioactivas, siendo
una estrategia prometedora para el descubri-
miento de especies químicas para el control
de agentes patógenos. Trabajos previos in-
dican la habilidad antagónica de Pseudovi-
brio denitrificans contra Vibrios patógenos
(7) lo que los convierte en una herramienta
útil contra vibriosis. A pesar de que se han
identificado compuestos antimicrobianos en
cepas de Pseudovibrios como el ácido tropo-
ditiético (TDA) (8) se desconoce la naturale-
za de este antagonismo.
Entre las técnicas que se emplean para el es-
tudio de sistemas biológicos, se encuentra la
metabolómica. Existen dos tipos de estudio
en metabolómica: (a) direccionados y (b) no
direccionados, la diferencia de estos radica
en que en metabolómica direccionada se rea-
liza el análisis de un grupo pequeño de me-
tabolitos, mientras en la metabolómica no
direccionada se analiza el metaboloma com-
pleto (9). Se define como metabolito a cual-
quier molécula de bajo peso molecular que
es encontrada en un sistema biológico (10). Uno de los enfoques
de la metabolómica nos permite comprender los cambios que
suceden en el metaboloma de un sistema biológico como con-
secuencia de estímulos específicos (11). Como ejemplo tenemos
el estudio de las interacciones de antagonismo, modificaciones
genéticas, estímulo fisiológico, entre otros (12).
Para el estudio del metaboloma la cromatografía líquida (LC)
es la opción preferencial ya que más del 90% de los metabolitos
son polares y no volátiles (13). Los sistemas de detección aco-
plados a los cromatógrafos se pueden basar en la medición de
la capacidad de una molécula para absorber energía lumínica
(espectrofotometría) o en la medición de la relación de su masa/
carga luego de ser ionizado (espectrometría). Entre los detecto-
res espectrofotométricos encontramos el de arreglo de diodos
(DAD). El conjunto de señales que llegan al DAD conforman
picos y el conjunto de picos es conocido como cromatograma.
La quimiometría se encarga del análisis, interpretación y visua-
lización de datos de origen químico. Con un alto número de
cromatogramas obtenidos, la comparación y discriminación de
picos de interés demandarían gran cantidad de tiempo, además
de tener un grado de subjetividad dependiente de la persona
que realiza esta tarea (14).
El uso de algoritmos no supervisados que puedan manejar
un gran número de variables y de muestras al mismo tiempo
han ayudado a solucionar los cuellos de botella en el manejo de
grandes cantidades de datos, ya que existe un análisis numérico
de todas las variables presentes (15). De esta manera, las señales
obtenidas por LC-DAD pueden ser sometidas a un pre-proce-
samiento para la corrección de ruido químico, seguido de un
análisis discriminante entre muestras y/o grupos, como es el
Análisis de Componentes Principales (PCA), Cuadrados Míni-
mos Parciales Discriminantes (PLS-DA), entre otros (16).
El objetivo de este trabajo fue implementar el uso de algoritmos
quimiométricos para la identificación de variaciones en el me-
taboloma del antagonismo microbiano entre Pseudovibrio de-
nitrificans y Vibrio harveyi. Estas variaciones pueden ser usadas
como herramienta para la búsqueda de moléculas con actividad
antimicrobiana. Este trabajo representa un estudio preliminar
cuyos resultados nos guiarán a la purificación y elucidación es-
tructural de metabolitos con posible actividad antimicrobiana
II. MATERIALES Y MÉTODOS
Inducción del antagonismo
La cepa de P. denitrificans fue obtenida de muestras de in-
vertebrados marinos sésiles, colectados en la Reserva Ma-

22
ISSN 2477-9105 Número 22 Vol. 2 (2019)
rina El Pelado, provincia de Santa Elena, Ecuador (-1,93514
S, -80,79939 O) en el extremo sur del Pacífico este tropical.
La cepa empleada de V. ha r ve yi, fue tomada del cepario del
Centro Nacional de Acuicultura e Investigaciones Marinas
(CENAIM-ESPOL).
Para inducir el efecto antagonista entre los microorganis-
mos, el cultivo de V. harveyi fue ajustado por densidad óptica
a 620 nm hasta una absorbancia de 0.6; equivalente a 1x10
8
células por mL
-1
. Se usó una solución de cloruro de sodio
al 2% (J.T. Baker, Estados Unidos) para hacer la suspensión
de bacterias. De esta suspensión bacteriana 200 μl fueron
inoculados por extensión en placas Petri que contenían agar
marino (Difco, Estados Unidos). La cepa de P. denitrificans
fue cultivada por 48h en placas de agar marino y recogida en
forma de cúmulo con ayuda de un asa.
Se inoculó el cúmulo formado de P. denitrificans como un
punto (de diámetro ~ 2-3 mm) en la superficie de la placa de
agar marino en la que previamente fue extendida la suspen-
sión de V. har ve y i para obtener el tratamiento de antagonis-
mo. Las placas con el inóculo y el cúmulo fueron incubadas
a 28 °C durante 72 horas en una incubadora (Heratherm®
IGS 180, Thermo Scientific, Alemania). Como controles se
cultivaron V. harveyi y P. denitrificans en placas separadas.
Se incluyeron placas con medio de cultivo sin inocular como
control negativo (blanco).
Preparación de la muestra para cromatografía líquida
Se recuperaron tanto las colonias bacterianas (suspensión de
bacterias) como el medio de cultivo utilizado. Las colonias
fueron suspendidas en agua grado I y transferidas a embu-
dos de separación. El medio de cultivo fue cortado en tro-
zos de aproximadamente 1 cm
3
para luego ser transferidos a
frascos con tapa rosca, los controles de bacterias cultivadas
por separado y el medio de cultivo sin inocular recibieron
el mismo tratamiento. Ambos tipos de muestra fueron so-
metidas a extracción con una parte de acetato de etilo grado
analítico (J.T. BAKER, Estados Unidos) por cada parte de
muestra. Se recuperó la fase orgánica y se evaporó el solven-
te en rotavapor (Buchi, RII, Suiza). Los residuos secos tanto
de la muestra de suspensión de bacterias y de agar fueron
sometidos a Extracción en Fase Sólida (SPE por sus siglas en
inglés) con cartuchos SPE C18 (Thermo Scientific, HYPER-
SEC, Estados Unidos), utilizando cinco diferentes eluyentes,
10 mL por cada uno: H
2
O (F1), H
2
O:MeOH 1:1 (F2), MeOH
(F3) (J.T. Baker, Trinidad y Tobago), MeOH:CH
2
CL
2
1:1 (F4),
y CH2CL2 (F5) (SupraSolv®, Merck, Alemania). La primera
y quinta fracción fueron almacenadas, las fracciones restan-
tes fueron evaporadas hasta sequedad.
Se recuperaron los residuos de SPE con el
mismo solvente usado en la elución para
cada fracción, se filtraron a través de fil-
tros de 0.22 µm y se inyectaron en el cro-
matógrafo líquido de ultra alta eficiencia
con detector de arreglo de diodos y de-
tector evaporativo de dispersión de luz
(UPLC-DAD-ELSD) (Waters, H-CLASS
1, Singapur). Las corridas cromatográfi-
cas fueron ejecutadas con una columna de
fase reversa (C
18
) de dimensiones 50 x 2.1
mm y de 1,7 µm de tamaño de partícula
(ACQUITY, Waters, Irlanda). La fase mó-
vil estuvo constituida por acetonitrilo (Sig-
ma-Aldrich, Corea del Sur) y agua grado I,
acidificados con 0.1% de ácido trifluoroa-
cético (Sigma-Aldrich, Francia). Se realizó
un gradiente de agua (A) y acetonitrilo (B)
de 0-2 minutos 10% B, de 2-10 minutos B
aumentó al 100%, de 10-12 minutos B se
mantuvo y finalmente de 12-15 minutos B
bajó al 10%. La absorbancia se registró a
254 nm.
Análisis de datos
Se tuvo un total de cuatro tratamientos (P.
denitrificans, V. h ar ve yi, antagonismo y
blanco), con dos niveles (agar y suspensión
de bacterias) con tres repeticiones cada
uno, de las que se analizaron las fracciones
F2, F3 y F4 por cada réplica, teniendo 72
cromatogramas como total. Los cromato-
gramas fueron exportados con el software
Empower (Waters, versión 3.3, Massachu-
set), e importados en el lenguaje estadísti-
co R versión 3.4 para análisis posteriores.
Todos los cromatogramas fueron recorta-
dos en los primeros 4 minutos. Después, se
corrigió la línea de base y se procedió con
el alineamiento de picos (17). Los datos
fueron sometidos a un escalamiento y cen-
trados en la media, como pretratamiento
antes de realizar el PCA.
III. RESULTADOS Y DISCUSIÓN
En la figura 1 se pueden observar los cro-
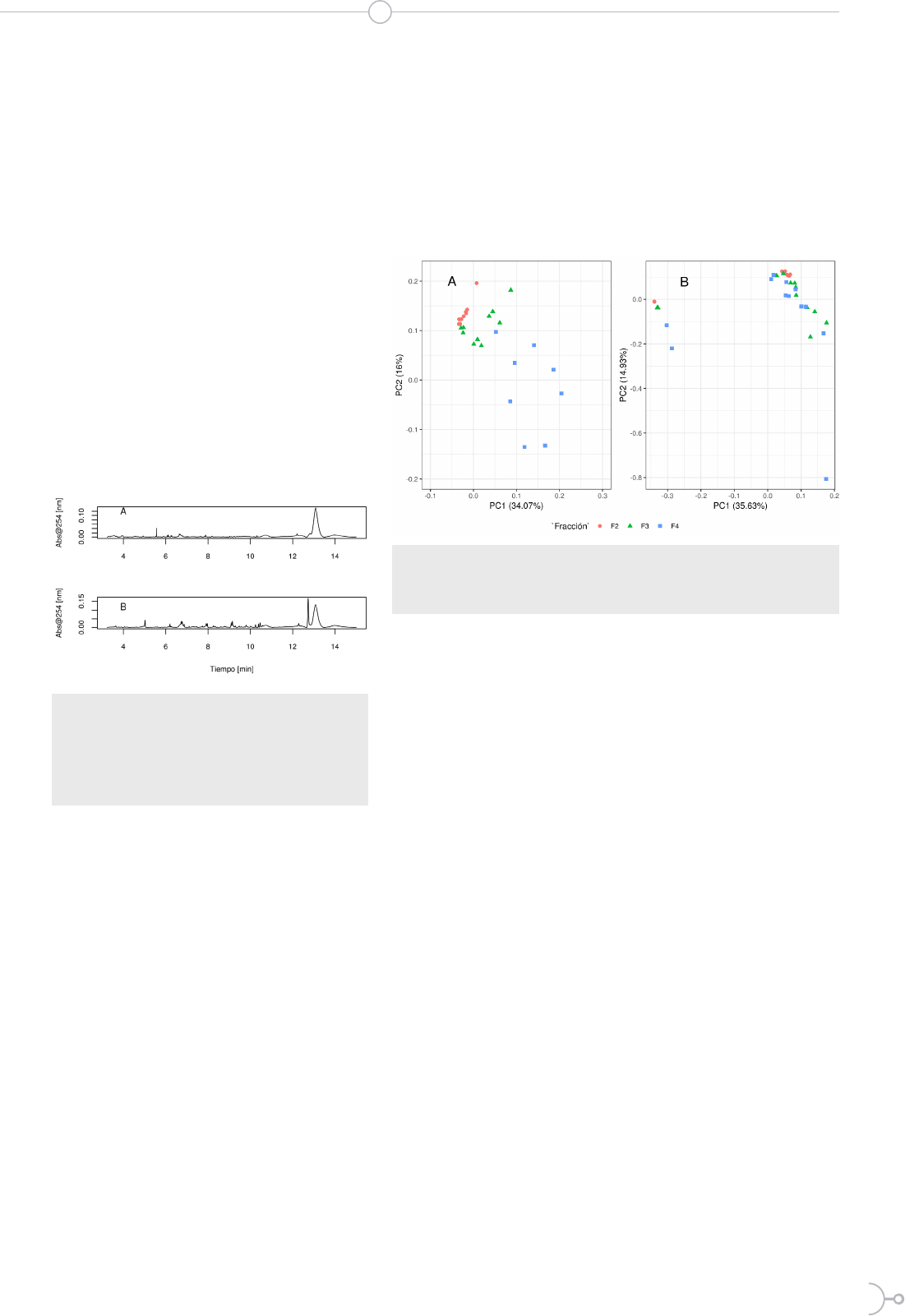
23
matogramas de la fracción F3 del agar
(Fig. 1A) y de la suspensión de bacterias
(Fig. 1B) de las muestras en las que se indu-
jo el antagonismo entre P. denitrificans y V.
harveyi. El análisis de reconocimiento de
patrones fue empleado para encontrar las
diferencias entre las cepas cultivadas indi-
vidualmente, el antagonismo entre ellas y
el blanco de agar (no inoculado). Una vez
corregida la línea base, se procedió con el
PCA con datos centrados en la media, y
escalados. El centrar en media corrige las
señales producidas por artefactos quími-
cos, como impurezas o cambios bruscos
de fase móvil. Mientras que el escalado
promueve que todas las variables tengan
un mismo factor de importancia.
En la figura 2 se pueden observar los re-
sultados del PCA, tanto para los cromato-
gramas del agar (Fig. 2A) como los croma-
togramas de las suspensiones de bacterias
(Fig. 2B).
Mediante la comparación de la dispersión
de los cromatogramas en el espacio pro-
yectado en los dos primeros Componentes
Principales (PC), se puede observar que las
muestras de agar (Fig. 2A) tienen una me-
jor separación entre grupos de fracciones
recolectadas por SPE en comparación con
la misma proyección para las muestras de
colonias (Fig. 2B). Esta mejor separación
en grupos para las muestras de agar indica
una mayor diversidad de compuestos pre-
sentes en el medio de cultivo en compara-
ción con los compuestos presentes en las
muestras de bacterias.
Análogamente, se cree que acciones de competencia y/o an-
tagonismo por sustrato en organismos de ambientes acuá-
ticos están relacionados con la secreción de compuestos de
carácter hostil que se difunden en el medio, ayudando a la
competencia con otros individuos (18).
Los cromatogramas de las fracciones F2 y F4 del medio de
cultivo presentan solapamiento y esparcimiento severo, res-
pectivamente (Fig. 2A); por lo que fueron descartados para
los análisis discriminantes posteriores para encontrar gru-
pos dentro de las fracciones. Por lo tanto, se realizó el PCA
solo para los picos de los cromatogramas de F3 de las mues-
tras de agar para los distintos tratamientos.
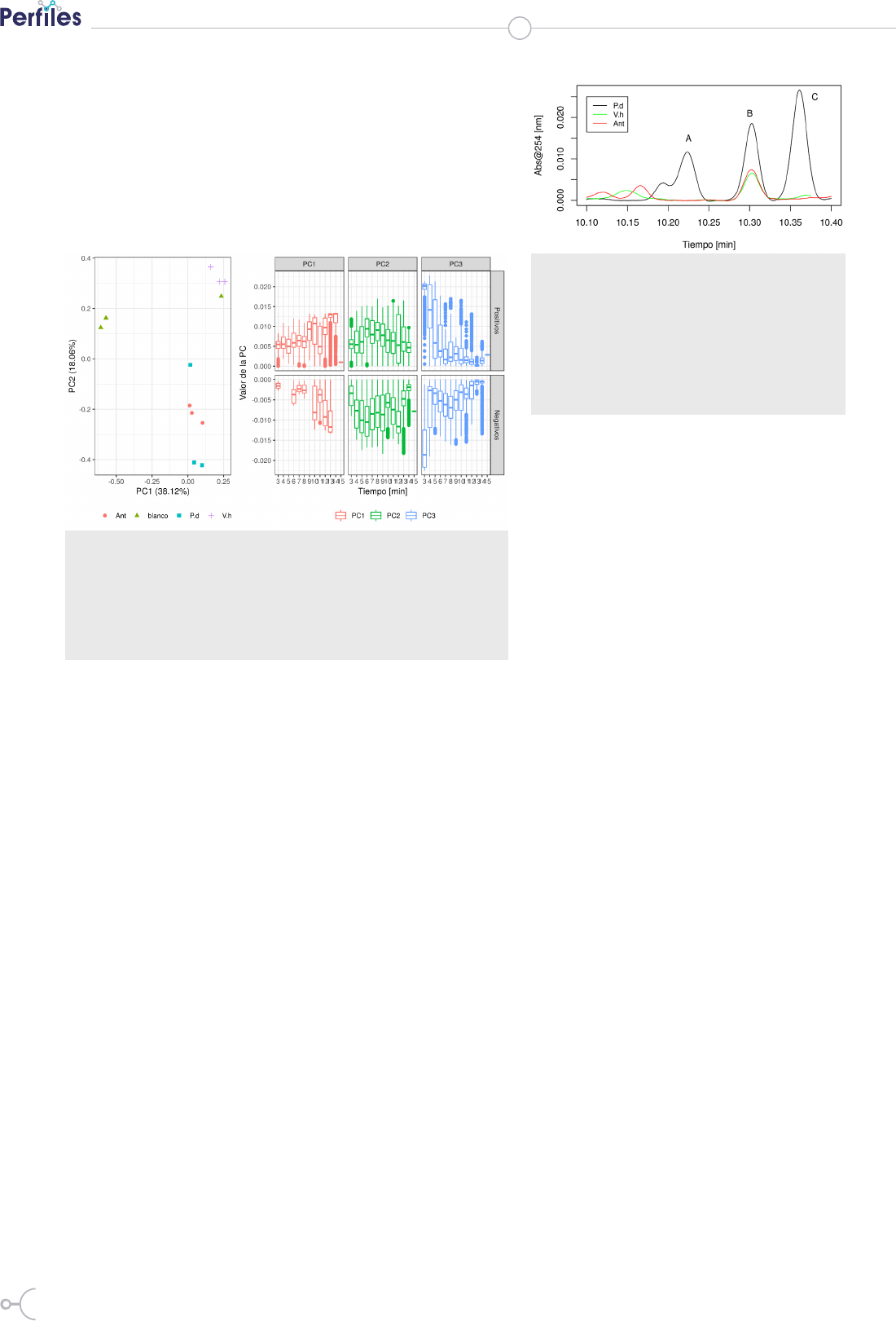
En la figura 3 se presentan los scores del análisis de los tra-
tamientos P. denitrificans (P.d.), V. ha r ve yi (V.h.), Antago-
nismo (Ant.) y Blanco (Fig. 3A), y también se presentan los
loadings (Fig. 3B). Por un lado, podemos analizar la disper-
sión de los cromatogramas para encontrar similitudes entre
ellos (Fig. 3A).
Existe una estrecha relación entre los cromatogramas de an-
tagonismo con los de P. denitrificans, por lo que se asume
que el metaboloma de estos dos grupos están más relaciona-
do entre sí, y son diferentes con el blanco y V. h ar ve yi. Ade-
más el blanco y V. har ve y i son diferentes entre sí, a pesar de
que una de las tres réplica del blanco se encuentra cercana a
V. harveyi, esto podría deberse a un artefacto en el procesa-
miento de las muestras.
En otras palabras, los tratamientos P. denitrificans y antago-
nismo presentan más compuestos similares entre si en algún
Chalén, Quiroz, Mogollon, Domínguez, Rodríguez
Figura 1. Cromatogramas representativos del antagonismo
entre Pseudovibrio denitricans y Vibrio harveyi. A es el cro-
matograma de la fracción F3 del agar y B es el cromatograma
de la fracción F3 de la suspensión de bacterias. La cromatogra-
fía fue realizada en fase reversa con gradiente, una columna
C18 y la absorbancia fue medida a 254 nm con un detector de
arreglo de diodos (DAD).
Figura 2. Análisis de Componentes Principales (PCA) de los cromatogramas de las frac-
ciones F2, F3 y F4 del agar (A) usado en el cultivo de las bacterias y de la suspensión de
bacterias (B) recuperadas luego del cultivo. Todos los cromatogramas fueron centrados
en media y escalados, con n = 36 para el agar y n = 36 para la suspensión de bacterias.
24
ISSN 2477-9105 Número 22 Vol. 2 (2019)
punto del cromatograma. Adicionalmente, con el objetivo
de encontrar los compuestos responsables del la clasificación
de las muestras, se analizaron los loadings negativos (Fig. 3B)
de los dos primeros PCs, encontrando que en ambos com-
ponentes, entre el minuto 10 y 11 se encuentran compuestos
que producen separación entre las muestras.
Se realizó una inspección supervisada de todos los cromato-
gramas basados en los menores loadings obtenidos (Fig. 4).
En base a la figura 3B se establece que en el minuto 10 exis-
ten compuestos cuya intensidad y/o presencia cambia entre
tratamientos. Los tres compuestos más variables se mues-
tran en la Figura 4 (A, B y C).
El compuesto A (Fig. 4) cambia levemente su tiempo de re-
tención entre tratamientos. El compuesto B tiene la misma
intensidad para V. har ve y i y antagonismo, sin embargo, la
intensidad para P. denitrificans es el doble, por lo que la inte-
racción de antagonismo suprime la producción de este com-
puesto en las cepas de P. denitrificans pero no en V. h ar vey i .
Adicionalmente, el pico C es producido únicamente en P.
denitrificans, y su ausencia en el tratamiento de antagonis-
mo sugiere que dicho compuesto ha sido metabolizado du-
rante la interacción bacteriana.
En otras palabras, es probable que el pico C sea un compues-
to que pertenezca al armamento químico de P. denitrificans
contra su antagonista. Una confirmación estructural de las
moléculas de interés representadas por los picos A, B y C
debe hacerse con el uso de herramientas de mayor poder
analítico tales como espectrometría de masas, espectrosco-
pía de infrarrojo y resonancia magnética nuclear.
IV. CONCLUSIONES
Este trabajo presenta la aplicación de algo-
ritmos quimiométricos en conjunto con la
cromatografía líquida de ultra alta eficiencia
acoplada a un detector de arreglo de diodos
para el entendimiento del antagonismo mi-
crobiano entre P. denitrificans y V. harveyi.
Con un análisis no supervisado como el PCA
se estableció el tipo de matriz con mayor va-
riabilidad de metabolitos entre tratamientos,
siendo el medio de cultivo.
Adicionalmente, el PCA de los cromatogra-
mas de la fracción F3 del medio de cultivo de-
terminó similitudes entre los tratamientos de
antagonismo y P. denitrificans, así como tam-
bién determinó diferencias del blanco con los
demás tratamientos. Mediante el análisis de
loadings se identificó tres entidades químicas
de importancia en esta relación de similitudes
y diferencias de la fracción F3 del agar.
La inspección supervisada de las tres enti-
dades químicas identificadas nos sugiere su
participación en el antagonismo, reduciendo
significativamente el rango de búsqueda para
análisis posteriores que determinarán la es-
tructura molecular de estas entidades.
De esta manera, el uso de la cromatografía lí-
quida de ultra alta eficiencia junto con algo-
ritmos quimiométricos demostró ser una he-
rramienta útil para el análisis y entendimiento
del metaboloma presente en el antagonismo
de P. denitrificans y V. harveyi facilitando el
tratamiento de una gran cantidad de datos y
reduciendo el tiempo necesario para análisis
posteriores.
Figura 4. Inspección de cromatogramas representativos en el
minuto 10. Se aprecian diferencias entre tres picos: A, B y C.
El pico A está presente en P. denitricans y en el Antagonismo
(con un leve cambio en el tiempo de retención). El pico B, pre-
senta la misma intensidad para V. harveyi y el Antagonismo,
pero tiene el doble de intensidad para P. denitricans por lo
que se presume que la interacción entre estas cepas induce la
supresión del pico B en P. denitricans. El pico C es observado
únicamente en P. denitricans sugiriendo que este compuesto
puede intervenir en la interacción con V. harveyi.
Figura 3. PCA de los cromatogramas de la fracción F3 del agar. En A se presentan los
scores del análisis de los tratamientos P. denitricans (P.d.), V. harveyi (V.h.), Antagonis-
mo (Ant.) y Blanco. En B se presentan los loadings negativos de los dos primeros Com-
ponentes Principales (PCs) con n = 12. Entre los dos primeros componentes se expresa
un total de 56.18% de la varianza de los datos. Por la disposición de los cromatogramas
en A, se puede inferir que el perl metabolómico del antagonismo es más parecido al
perl de P. denitricans. Además, en B es apreciable que el minuto 10 alberga los picos de
mayor importancia discriminante.

R
eferencias
25
V. AGR A DECIMIENTOS
Este trabajo fue financiado por la Secretaría
de Educación Superior, Ciencia, Tecnología
e Innovación (SENESCYT) en el marco del
proyecto "Caracterización de la biodiversidad
microbiológica y de invertebrados de la reser-
va marina El Pelado a escalas taxonómica, metabolómica y meta-
genómica, para uso en salud humana y animal" con permiso de
investigación 005-17 IC-FAU-DPSE/MA y contrato de acceso a
recurso genético MAE-DNB-CM-2015-0021. Parte de esta inves-
tigación se realizó en el marco del programa conjunto de postgra-
dos VLIR NETWORK Ecuador.
1. Burgess JG, Jordan EM, Bregu M, Mearns-Spragg A, Boyd KG. Microbial antagonism: a neglected ave-
nue of natural products research. Journal of Biotechnology 70(1-3):27-32. [Archidoris pseudoargus p. 30. J
Biotechnol 70. 1999;70:27–32.
2. Rypien KL, Ward JR, Azam F. Antagonistic interactions among coral-associated bacteria. Environ Micro-
biol. 2010;12(1):28–39.
3. You THC, Feng LZC, Kong JWF. Biocontrol potential of antagonist Bacillus subtilis Tpb55 against tobac-
co black shank. BioControl. 2015;
4. Spadaro D, Droby S. Trends in Food Science & Technology Development of biocontrol products for
postharvest diseases of fruit : e importance of elucidating the mechanisms of action of yeast antago-
nists. Trends Food Sci Technol [Internet]. 2016;47:39–49. Available from: http://dx.doi.org/10.1016/j.
tifs.2015.11.003
5. Palazzini J, Roncallo P, Cantoro R, Yerkovich N, Echenique V, Torres A, et al. Reduction of Deoxynivale-
nol Accumulation and Phytohormone Induction by Two Selected Antagonists. Toxins (Basel). 2018;10(88).
6. Khabbaz SE, Zhang L, Cáceres LA, Sumarah M, Wang A, Abbasi PA. Characterisation of antagonistic
Bacillus and Pseudomonas strains for biocontrol potential and suppression of damping-o and root rot
diseases. Ann Appl Biol. 2015;166(3):1–16.
7. Raina J-B, Tapiolas D, Motti CA, Foret S, Seemann T, Tebben J, et al. Isolation of an antimicrobial com-
pound produced by bacteria associated with reef-building corals. PeerJ. 2016;4:e2275.
8. Penesyan A, Tebben J, Lee M, omas T, Kjelleberg S, Harder T, et al. Identication of the Antibacterial
Compound Produced by the Marine Epiphytic Bacterium Pseudovibrio sp. D323 and Related Sponge-As-
sociated Bacteria. Mar Drugs. 2011 Aug;9(8):1391–402.
9. Murray JA. Qualitative and quantitative approaches in comprehensive two-dimensional gas chroma-
tography. J Chromatogr A [Internet]. 2012;1261:58–68. Available from: http://dx.doi.org/10.1016/j.chro-
ma.2012.05.012
10. Patti G, Yanes O, Siuzdak G. Metabolomics: e apogee of the omics trilogy. Int J Pharm Pharm Sci.
2012;5(SUPPL. 2):45–8.
11. Madsen R, Lundstedt T, Trygg J. Chemometrics in metabolomics-A review in human disease diagnosis.
Anal Chim Acta. 2010;659(1–2):23–33.
12. Fiehn O. Metabolomics--the link between genotypes and phenotypes. Plant Mol Biol [Internet].
2002;48(1–2):155–71. Available from: http://www.ncbi.nlm.nih.gov/pubmed/11860207
13. eodoridis GA, Gika HG, Want EJ, Wilson ID. Liquid chromatography-mass spectrometry based glo-
bal metabolite proling: A review. Anal Chim Acta [Internet]. 2012;711:7–16. Available from: http://dx.doi.
org/10.1016/j.aca.2011.09.042
14. Fraga CG, Prazen BJ, Synovec RE. Objective data alignment and chemometric analysis of compre-
hensive two-dimensional separations with run-to-run peak shiing on both dimensions. Anal Chem.
2001;73(24):5833–40.
15. Szymańska E. Modern data science for analytical chemical data – A comprehensive review. Anal Chim
Acta. 2018;1028.
16. Bartel J, Krumsiek J, eis FJ. Statistical Methods for the Analysis of High-roughput Metabolomics
Data. Comput Struct Biotechnol J [Internet]. 2013;4(5):e201301009. Available from: http://linkinghub.else-
vier.com/retrieve/pii/S2001037014600520
17. Wehrens R, Bloemberg TG, Eilers PHC. Fast parametric time warping of peak lists. Bioinformatics.
2015;31(18):3063–5.
18. Mille-Lindblom C, Fischer H, Tranvik LJ. Antagonism between bacteria and fungi: Substrate competition
and a possible tradeo between fungal growth and tolerance towards bacteria. Oikos. 2006;113(2):233–42.
Chalén, Quiroz, Mogollon, Domínguez, Rodríguez